近期,谭雅岚博士与武汉大学谭志杰教授等合作在RNA三维结构评估方面取得了重要进展,相关成果以“rsRNASP: A residue-separation-based statistical potential for RNA 3D structure evaluation”为题在该领域国际权威期刊Biophysical Journal上发表,该论文被选为当期的New and Notable论文,并由北卡罗来纳大学的Laederach教授等以“A novel algorithm for ranking RNA structure candidates”为题发表评述。该文的第一单位为武汉纺织大学非线性科学研究中心,第一作者为数理学院谭雅岚博士。Biophysical Journal为美国生物物理学会会刊,由Cell出版社出版。
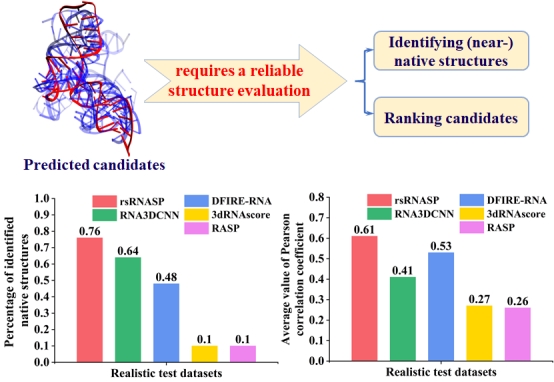
RNA除了在序列层面存储和传递遗传信息外,其中非编码RNA还有着其它诸多重要生物功能,包括基因调控和催化等。RNA的功能与其结构紧密相关,因此获得RNA的高精度三维结构对理解其生物功能至关重要。不管是RNA三维结构的实验测定还是理论预测,往往会产生具有很多个可能候选结构的结构集,因而对候选结构进行结构评估是获得高精度三维结构的关键步骤之一。
现有用于RNA三维结构评估的统计势对来源于结构预测的实际测试集效果不佳。考虑到RNA三维结构折叠通常是逐级进行的因而不同碱基分离度范围内的相互作用在RNA的折叠中可能扮演着不同角色,谭雅岚博士等提出了一个新的基于碱基分离度的RNA三维结构评估统计势,并命名为rsRNASP。通过广泛的测试发现,对来源于结构预测模型的实际测试集,rsRNASP在天然态结构进行识别、构象集排序等评估指标上都明显优于现有传统统计势(包括DFIRE-RNA、3dRNAscore以及RASP)和基于神经网络训练得到的统计势(包括3DRNACNN和ARES)。此外,论文还分析了rsRNASP捕获的RNA三维结构中的重要物理相互作用,从而揭示了其良好性能的微观机制。rsRNASP的提出为得到高精度RNA三维结构提供了新的工具,rsRNASP及其相关数据库已开源供同行使用。
论文链接:https://www.cell.com/biophysj/fulltext/S0006-3495(21)00985-1
New and Notable(北卡罗来纳大学Laederach教授等评述)链接:https://www.cell.com/biophysj/fulltext/S0006-3495(21)03895-9

